분산처리를 잘 사용해 볼 수 있는 예제가 없을까 하다가 주변 지인의 추천으로 유전알고리즘을 적용해 보기로 했다.
1. 간단한 유전알고리즘 코드 짜기
간단한 유전 알고리즘의 코드를 짜보자.
유전알고리즘은 난수로 생성된 인구에 약간의 확률로 변이를 주고
두 인구를 융합함으로 2세대를 만들어내는 작업을 반복한다.
멀티 프로세싱에 아주 적합한 예시라고 할 수 있다.
import random
def init_population(dna_length, population):
return ["".join(random.choice("01") for _ in range(dna_length)) for _ in range(population)]
def fitness(dna):
return dna.count("1")
def select(dnas, population):
sorted_pop = sorted(
dnas,
key=fitness,
reverse=True,
)
return sorted_pop[:population//2]
def crossover(dna1, dna2, crossover_rate, dna_length):
if random.random() < crossover_rate:
pivot = random.randint(0, dna_length - 1)
return dna1[:pivot] + dna2[pivot:], dna1[pivot:] + dna2[:pivot]
else:
return dna1, dna2
def mutate(dna, mutation_rate):
result = ""
for gene in dna:
if random.random() < mutation_rate:
gene = abs(int(gene)-1)
result = f"{result}{gene}"
return result위와 같이
- 인구를 초기화 하는 함수
- 적합도 계산 함수
- 적합도가 높은 순서대로 절반의 인구를 취하는 함수
- 교배 함수
- 돌연변이 함수
를 짜준다.
import utils.tools as T
POPULATION = 100
DNA_LENGTH = 10
MUTATION_RATE = 0.01
CROSSOVER_RATE = 0.7
GENERATIONS = 100
dnas = T.init_population(DNA_LENGTH, POPULATION)
for _ in range(GENERATIONS):
generation = []
for dna1, dna2 in zip(dnas[0::2], dnas[1::2]):
dna1_mutant = T.mutate(dna1, MUTATION_RATE)
dna2_mutant = T.mutate(dna2, MUTATION_RATE)
children = T.crossover(dna1_mutant, dna2_mutant, CROSSOVER_RATE, DNA_LENGTH)
generation.extend(children)
dnas = T.select(generation, POPULATION)
print(dnas)이제 그 함수들을 불러와서 간단한 유전 알고리즘을 진행한다.
1의 수가 많을 수록 적합도가 높으므로 1이 많은 dna가 가장 먼저 나타날 것이다.

위와 같은 결과를 얻었다.
2. MPI 적용하기
이걸 단순히 파이썬 multiprocessing 으로 처리할 수도 있지만 한대의 노드에서만 진행할 수 있다.
대신에 여러대의 노드가 있으면 그만큼 더 효율적으로 진행할 수 있기 때문에 멀티노드로 진행해 본다.
우선은 유의미한 시간이 걸릴만큼 파라미터를 늘려준다.
import utils.tools as T
POPULATION = 1000
DNA_LENGTH = 100
MUTATION_RATE = 0.01
CROSSOVER_RATE = 0.7
GENERATIONS = 2000
tic = time()
dnas = T.init_population(DNA_LENGTH, POPULATION)
for _ in range(GENERATIONS):
generation = []
for dna1, dna2 in zip(dnas[0::2], dnas[1::2]):
dna1_mutant = T.mutate(dna1, MUTATION_RATE)
dna2_mutant = T.mutate(dna2, MUTATION_RATE)
children = T.crossover(dna1_mutant, dna2_mutant, CROSSOVER_RATE, DNA_LENGTH)
generation.extend(children)
dnas = T.select(generation, POPULATION)
toc = time()
print(toc - tic)이정도로 늘려주니 라즈베리파이에서 55초정도 나왔다.
이제 이 파일을 복사해서 mpi를 사용할 수 있게 바꿔보자.
from time import time
from functools import reduce
from mpi4py import MPI
import utils.tools as T
comm = MPI.COMM_WORLD
rank = comm.Get_rank()
world_size = comm.Get_size()
POPULATION = 1000
DNA_LENGTH = 100
MUTATION_RATE = 0.01
CROSSOVER_RATE = 0.7
GENERATIONS = 2000
if rank == 0:
tic = time()
dnas = T.init_population(DNA_LENGTH, POPULATION)
for _ in range(GENERATIONS):
local_dnas_size = POPULATION // world_size
local_dnas = comm.scatter([
dnas[i*local_dnas_size:(i+1)*local_dnas_size] for i in range(world_size)
], root=0)
generation = []
for dna1, dna2 in zip(local_dnas[0::2], local_dnas[1::2]):
dna1_mutant = T.mutate(dna1, MUTATION_RATE)
dna2_mutant = T.mutate(dna2, MUTATION_RATE)
children = T.crossover(dna1_mutant, dna2_mutant, CROSSOVER_RATE, DNA_LENGTH)
generation.extend(children)
all_generation = comm.gather(generation, root=0)
if rank == 0:
generation = reduce(lambda x, y: x + y, all_generation)
dnas = T.select(generation, POPULATION)
if rank == 0:
print(f"best : {dnas[:2]}\nworst : {dnas[-2:]}")
toc = time()
print(toc - tic)
멀티프로세싱을 작업할 때는 전략을 잘 짜야 한다.
우선 서로 다른 자원에 동일한 연산을 취해야할 때 가장 효과가 좋다.
즉 점화식으로 표현되지 않는 모든 루프 연산에 대해서 취해주는 것이 좋다.
나는 여기서 dna를 나누고 각 노드에 뿌려줌으로써 멀티프로세싱을 구현하였다.

싱글노드는 여전히 54초 정도 나온다.
같은 노드의 4개 코어를 사용하면

32초, 대략 40% 정도 줄었다.
이번엔 서로 다른 노드의 4개 코어를 사용하면
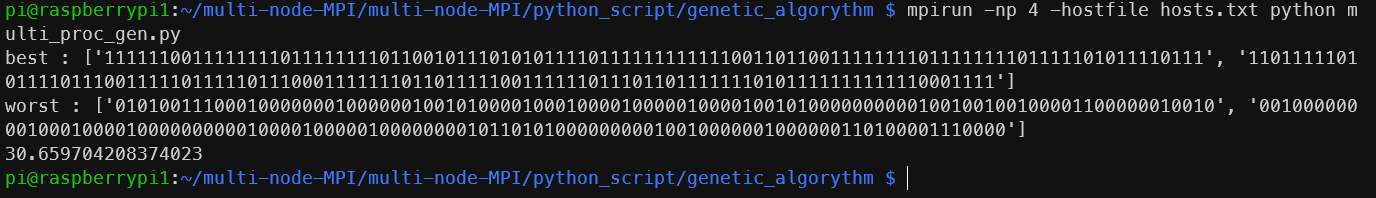
유의미한 변화는 없다.
이번에는 모든 노드의 모든 코어를 사용하면
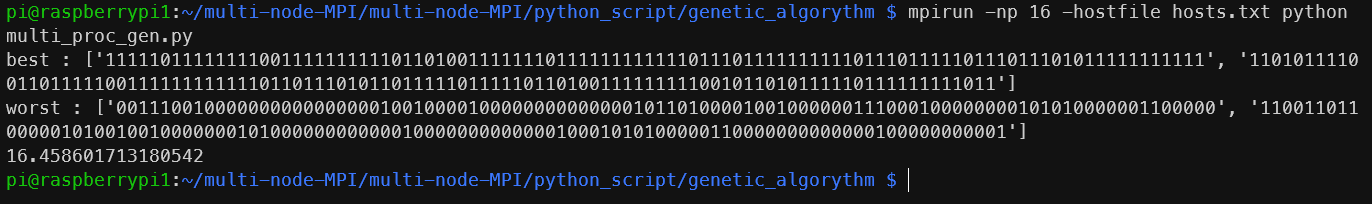
16초까지 줄어드는 것을 볼 수 있다.
comm.scatter 를 통해서 값을 덮어쓰고
연산을 각자 진행한 뒤에
comm.gather 를 통해서 값을 하나로 모으는 것이 핵심이다.
다음은 이 모든 과정을 쿠버네티스에 어떻게 녹일 수 있을지 고민해 보겠다.
'Backend MLOps > On-premise setting' 카테고리의 다른 글
| [k8s] Jetson Nano 병렬처리 해보기 (1) | 2023.10.12 |
|---|---|
| [k8s] Jetson Nano에 k3s 설치하고 워커노드로 구축하기 (0) | 2023.09.24 |
| [MPI] 파이썬으로 멀티노드 분산컴퓨팅 처리 (0) | 2023.09.12 |
| [MPI] 멀티노드로 병렬연산, 분산컴퓨팅 처리하기 (0) | 2023.09.03 |
| [k8s] NFS 기반 PersistentVolume 직접 구축하기 - 2 (0) | 2023.09.02 |




댓글